上海金畔生物科技有限公司代理New England Biolabs(NEB)酶试剂全线产品,欢迎访问官网了解更多产品信息和订购。
NEBNext®FFPE DNA 修复模块 v2 收藏
Download:
- isoschizomers |
- compatible ends |
- single letter code
产品特点
该新产品进一步提升了二代测序(NGS)流程中 FFPE DNA 的修复效果
概述
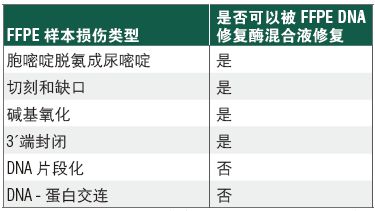
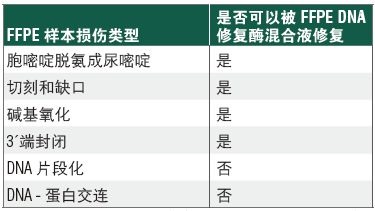
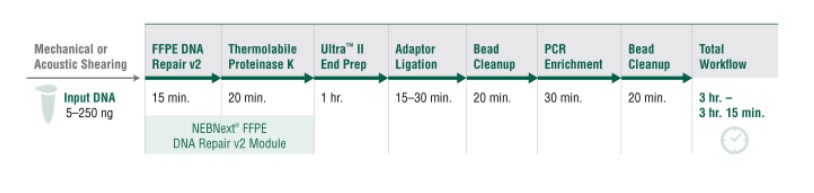
FFPE 是福尔马林固定石蜡包埋样本,由于其固定和保存的方法,给其中的 DNA 造成严重损伤,因此从 FFPE 样本中获取高质量的序列数据就变得具有挑战性。NEBNext® FFPE DNA 修复模块 v2 是一种优化的酶混合物,旨在修复 FFPE 样本 DNA,同时配有经过优化的试剂,能实现更精简的 NGS 文库制备流程。
产品描述
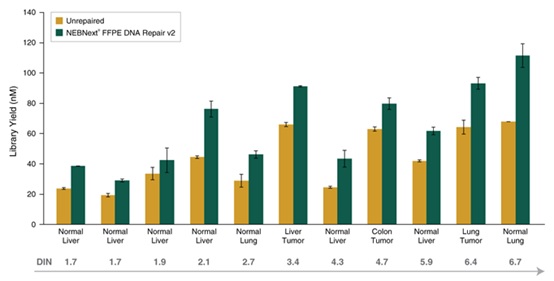
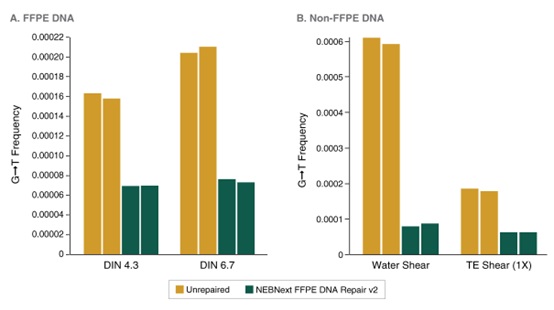
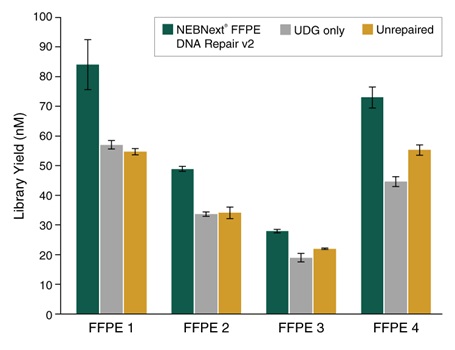
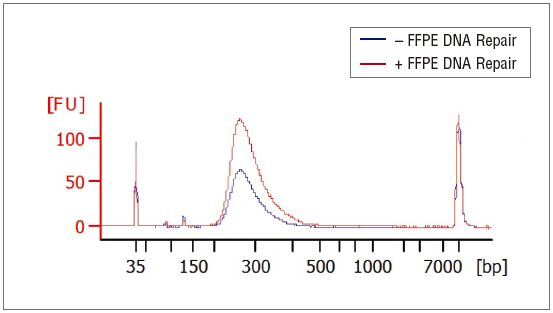
NEBNext® FFPE DNA 修复模块 v2 修复不同质量的 FFPE DNA 后,都能进行高效的文库制备。选用 25 ng 不同质量和组织来源的 Covaris® 超声打断后的 FFPE DNA 样本制备文库。使用 NEBNext® FFPE DNA 修复模块 v2 修复后使用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)建库,共进行 9 个 PCR 循环。使用 Agilent® HS D1000 TapeStation® 进行文库定量。NEBNext® FFPE DNA 修复模块 v2 根据起始 DNA 的质量和损伤类型,不同程度地提高 FFPE 文库的产量。误差条表示每个文库样本两次重复的标准差。
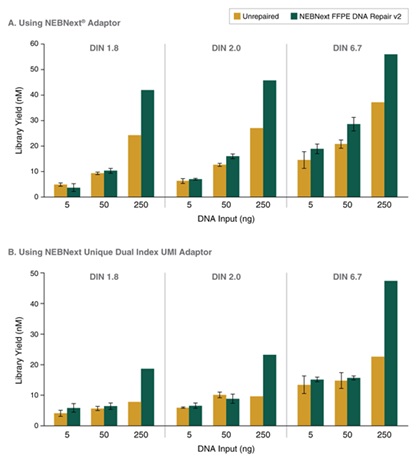
使用 NEBNext® 发夹结构接头和 Unique 双端 Index 引物,UMI 接头,NEBNext® FFPE DNA 修复模块 v2 修复 5 – 250 ng FFPE DNA 后,都能进行高效的文库制备。选用 5 ng、50 ng 和 250 ng 三种不同质量的正常肝脏 FFPE DNA 样本制备文库,比较使用和不使用 NEBNext® FFPE DNA 修复模块 v2 修复的文库制备效果。使用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)和(a)NEBNext® 发夹结构接头,针对 5 ng、50 ng 和 250 ng 三种起始 DNA 分别使用 11、8 和 6 个 PCR 循环制备文库,以及(b)NEBNext® Unique 双端 Index 引物,UMI 接头(NEB #E7395),针对 5 ng、50 ng 和 250 ng 三种起始 DNA 分别使用 11、8 和 6 个 PCR 循环制备文库。使用 Agilent® HS D1000 TapeStation® 进行文库定量,图中绘制了 2 个文库(5 和 50 ng)和 1 个重复(250 ng)的平均产量。误差条表示 5 和 50 ng 文库重复的标准差。使用 NEBNext® 发夹结构接头和 Unique 双端 Index 引物,UMI 接头,针对 5 ng 至 250 ng 的 FFPE DNA 样本,NEBNext® FFPE DNA 修复模块 v2 都能进行高效的文库制备。
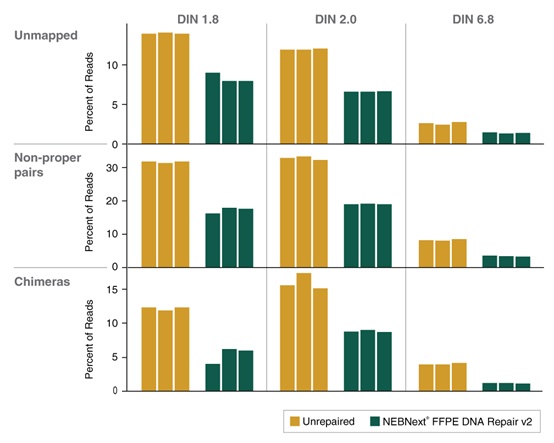
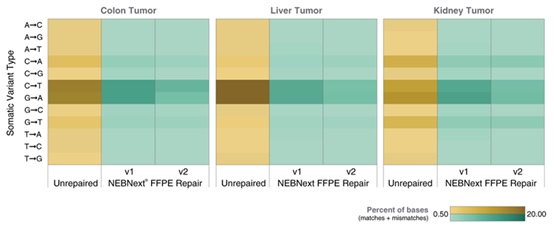
NEBNext® FFPE DNA 修复模块 v2 能提高各项文库质量指标,包括比对率、正确匹配的双端 reads 和嵌合序列。选用三份 50 ng 不同质量的正常肝脏 FFPE DNA 样本(使用或未使用 NEBNext® FFPE DNA 修复模块 v2 修复),采用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)制备文库。使用 Illumina NextSeq® 500 测序。向下抽样 100 万个双端 Reads,并使用 Bowtie2(v2.3.2)将 Reads 与 GRCh38 人类参考基因组(RefSeq 884148)进行比对。使用 MarkDuplicates(v1.56.0)分析比对上的 Reads,使用 Picard SAM/BAM 比对汇总指标(v1.56.0)。使用 NEBNext® FFPE DNA 修复模块 v2 可提高比对率,并降低非正确匹配和嵌合序列。
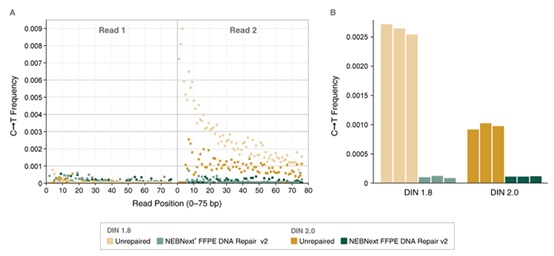
NEBNext® FFPE DNA 修复模块 v2 能修复 FFPE DNA 样本中存在的大量胞嘧啶脱氨损伤。选用两种 50 ng 不同质量的正常肝脏 FFPE DNA 样本(DIN 2.0 和 DIN 1.8,),使用或未使用 NEBNext® FFPE DNA 修复模块 v2 修复后,采用 NEBNext® Ultra™ II DNA 文库制备试剂盒(NEB #E7645)制备文库后,使用 Illumina NextSeq® 500 测序。向下抽样 100 万个双端 Reads,并使用 Bowtie2(v2.3.2)将 Reads 与 GRCh38 人类参考基因组(RefSeq 884148)进行比对。根据等式MAX([C T]-[G A])/([NC]+[NG]),EXP(-10)),使用 MarkDuplicates(v1.56.0)分析比对上的 Reads,使用 Tasmanian(V0.1.3)分析 dCdT(CT)突变。(a):在 Read 1 和 Read 2 中,绘制了 CT 突变频率与 Read 位置(0 – 75 bp)的函数关系图。当使用 NEBNext® FFPE DNA 修复模块 v2 修复后,在两个不同的 FFPE DNA 样本中,这些 CT 突变的丰度和位置偏差都显著降低。(b):Read 2 中的错误突变被量化为总频率。图中结果是每种情况下的两个重复数据。